5.2 dplyr::
Los principales cinco verbos (funciones) de dplyr son:
select filter mutate summarise arrange
select
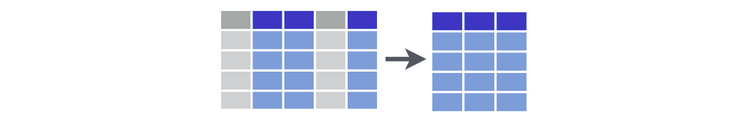
Vamos a seleccionar las variables: study, year y fungic del archivo soja:
soja_long %>%
select(trt, yield)
# Una selección “negativa” de las variables no deseadas daría un mismo resultado:
# soja_long %>%
# select(-bk)5.2.1 filter
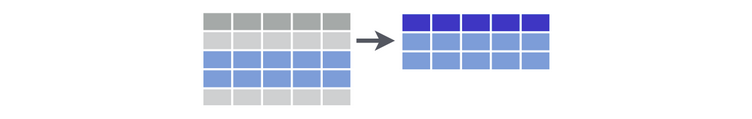
Semejante a subset. Condiciones separadas por comas equivalen a & de subset.
Filtremos la variable fungicida (fungic) por el testigo (ZZ_CHECK):
Ahora, agreguemos el trt A al dataset filtrado:
mutate
Permite operar sobre las columnas, modificando su naturaleza o haciendo operaciones sobre ellas (incluso generando nuevas columnas a partir de pre-existentes).
- Creación de nuevas variables (a partir de las existentes)
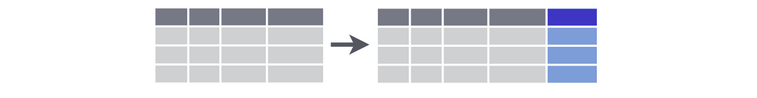
Supongamos que queremos analizar a yield en escala de quintal (100kg = 1qq)
- Conversión de tipos de variables:
Ahora que hemos re-organizado los datos, queremos chequear los tipos de variables que tiene el dataset:
5.2.2 summarise
Generalmente acompañada de la función group_by la cual permite aplicar un cálculo a las observaciones agrupando por niveles de algún factor (equivale a una tabla dinámica de Excel)

Veamos cuánto fue el rendimiento promedio y el desvío standard para cada fungicida a través de todos los ensayos:
Calculen el rendimiento mínimo y máximo de cada tratamiento
arrange
Ordenar columnas
- Función
subset
Filtremos a la variable Species reteniendo sólo a “setosa”
Filtremos a la variable Species reteniendo sólo a “setosa” + “virginica”
Anteponiendo el ! a la condición estaremos aplicando la negativa de la condición.
regenere
iris_set.virginicausando!para mejorar el código
Agreguemos una condición: a lo anterior quedémonos con aquellas filas en que Sepal.Length > 5
¿Qué pasa si cambiamos el operador
&por|?
iris4no es un codigo suscinto… reformulelo en iris5 para queidentical(iris4, iris5) == TRUE